本文来自微信公众号「果壳」(ID:Guokr42),作者核桃苗,爱范儿经授权发布。
只要你有一台能上网的电脑,你也可以为抗击疫情提供帮助。
下面这个动图,模拟了新冠病毒的刺突糖蛋白在真实世界中不断变化的样子。这个蛋白非常重要,是病毒入侵细胞的关键。这一成果,将为抗病毒药物的开发提供指导。
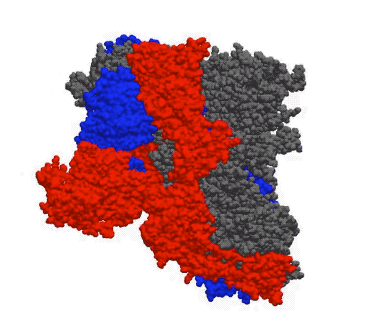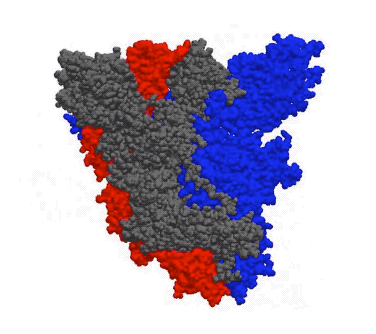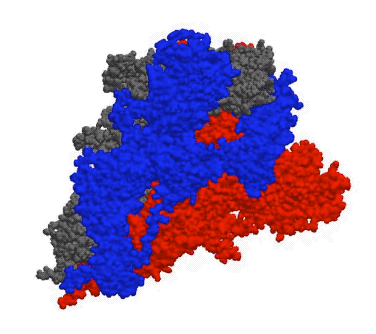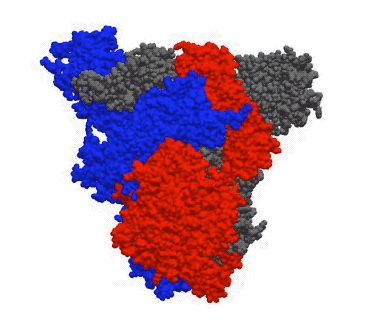
▲ 新冠病毒刺突糖蛋白的动态模拟。新冠病毒通过刺突糖蛋白与人体细胞上的 ACE2 受体蛋白结合,从而进入人体细胞 | Twitter:@Greg Bowman
你也许会认为,这是科学家们用庞大而昂贵的科学仪器生成的;但实际上,这个结果来自数十万普通人的个人电脑。这个项目叫做 Folding@home(直接翻译过来是 「在家进行蛋白折叠」),你也可以参与其中。
20 世纪 60 年代,科学家们开始尝试用射电望远镜侦听来自太空的无线电信号,寻找外星智慧生命的痕迹。分析这些数据需要巨大的运算能力,随着项目规模的扩大,分析所需的计算资源成了瓶颈。于是在千禧年来临前夕,当互联网正向世界的各个角落扩张的时候,他们产生了一个想法 —— 集合各家各户的电脑,共同完成计算任务。

▲ SETI 创立人法兰克・德雷克(Frank Drake)正在浏览射电望远镜收集到的数据 | Lou Dematteis / Reuters
1999 年 5 月 17 日,SETI@home(Search for ExtraTerrestrial Intelligence at Home,「在家搜寻外星智慧」)正式上线。这个项目由加州大学伯克利分校的程序员和天文学家共同运行,是最早的 「 志愿分布式计算项目」 之一。参与这个项目的人,不必把电脑邮寄给大学,只需要下载客户端。配置完毕后,电脑进入屏幕保护状态时,就会自动从服务器下载地外无线信号记录,并从中寻觅来自外星生命的问候。
现在个人电脑的速度比当时快了超过 100 倍,即便随时进行这些计算任务,也不会拖慢电脑的运行速度。因而,现在的项目也不像最初 SETI@Home 那样仅局限于屏幕保护时才运行,而是将何时运行的选择权交给了用户。用户可以选择在其他应用不那么活跃时才运行这个项目,或者设置成一边做其他事情一边运行。
「志愿分布式计算」 可以满足大规模科学计算的需求,它通过互联网,将个人计算机的运算能力集合起来。庞大的计算任务在服务器端被分割成许多小份的数据包,通过互联网分发给个人电脑;个人电脑分别计算完毕,再回传给服务器进行整合。你可以将 「分布式计算」 理解成一台特殊的超级计算机 —— 传统的超级计算机是在一个巨大的机房中,将大量处理器通过线缆连接起来,共同完成计算任务;而志愿分布式计算则是通过互联网,将世界各地的处理器连接起来进行计算。
▲ SETI@home 项目已于今年 3 月 31 日无限期休止,不过官方仍然鼓励大家加入其他的分布式计算项目,例如 COVID-19 的研究 | setiathome.berkeley.edu
在 SETI@home 开始后的二十年中,这一思路在各个领域得到应用。如今,在伯克利大学网络计算开放平台(BOINC)上,可以找到用于分析大型强子对撞机数据的 LHC@home、搜索脉冲星引力波的 Einstein@home、寻找大质数的 PrimeGrid 等各个学科的项目。而独立平台也有许多,例如用于检测地震的捕震网(Quake-Catcher Network),以及文章开头提到的专注于蛋白质折叠与动态的 Folding@Home。当初开发 SETI@home 的科学家大约没想到,20 年后,这项技术将被用于对抗一场全球瘟疫。在你阅读这篇文章的时候,Folding@home 正在将大量新冠病毒的分析任务,拆解分配给世界各地的计算机,为抗击疫情提供帮助。
SETI@home 上线一年半后,当上百万志愿者的电脑正在聆听星空的声音、寻找地外生命时,Folding@home 发布了。科学家们希望借助分布式计算的力量,模拟蛋白质折叠,进而了解它们在疾病中的作用,为寻找新的药物提供信息。过去的二十年中,这个项目为解析乳腺癌相关的异常激酶、寻找埃博拉病毒的潜在药物靶点、预测阿尔茨海默症中异常蛋白结构等一系列工作提供了帮助。在项目运行的第 7100 天、多灾多难的 2020 年的 2 月即将结束的时候,作为团队的领导,生物物理学家格雷格・鲍曼(Greg Bowman)在项目官网宣布,他们已经开始将 Folding@home 投入新冠病毒蛋白相关的工作,随后还提高了新冠病毒蛋白相关计算任务的优先级。
▲ Folding@home 团队 | foldingathome.org
蛋白质的基本单位是氨基酸,但氨基酸排列成长链之后,还需要通过折叠形成三维结构,才会发挥作用。折叠的过程、最终的结构以及折叠后蛋白的动态,都影响着蛋白质的功能,进而影响生命活动。科学家们有许多方法解析蛋白质的静态结构,例如冷冻电镜技术已经展示了新冠病毒的刺突糖蛋白结构。然而,这些方法只能显示蛋白质在特定瞬间的形象。鲍曼比喻道:「实验方法捕捉的蛋白结构就像橄榄球员们蓄势待发时的照片,能让我们看到阵型安排,但是没法看到整场比赛。」
▲ 蛋白质的静态结构就像球赛照片,能看到阵型安排,却看不到正常比赛 | foldingathome.org
不过,以这些静态形象为起点、展开计算机模拟,则可以预测蛋白质中每个原子的运动,展示出可以改变形态的区域,让科学家们看到整场比赛,进而针对性地制定攻防策略。以埃博拉病毒为例,过去人们认为其中的 VP35 蛋白没有可供药物作用的位点,但不久前计算机模拟发现,动态变化中产生的新结构是可以接受药物作用的 [1],这为寻找疗法提示了新方向。类似地,开发对抗新冠病毒的药物时,或许也能这样的研究里找到思路。唯一的问题是,这样的模拟需要巨大而昂贵的计算资源,生物学家办公室里的计算机,可能要不眠不休地工作上百年甚至上万年,才能模拟一个蛋白的动态。
好在全球有数十亿台个人电脑,热心地加入分布式计算网络的普通人,让模拟蛋白质动态所需的时间变得可以接受。时间进入 3 月,Folding@home 项目组在社交媒体上发出号召后,许多人加入了这个分布式计算网络。参与的用户数量从 2 月份的约 3 万人,增加至如今超过百万。「也许大家注意到安装时的卡顿了,」 鲍曼在社交网络上说,「有太多人在下载客户端了,服务器有点跟不上。我们正在努力让新服务器上线。」
▲ 许多 PC 硬件、电子游戏相关的厂商和媒体也呼吁用户加入其中 | Twitter:@PC MASTER RACE
虽然每台个人设备的运算能力微不足道,但是巨大的数量相加,使得可以利用的空闲算力超过了所有人的预期。目前世界上性能最强的超级计算机是 IBM 公司 「顶点」(Summit),每秒可以计算 20 亿亿次(每秒 2×10^17 次)。3 月 21 日,鲍曼在社交媒体上宣布,Folding@home 的总计算速度达到了 「顶点」 超级计算机的两倍。仅仅 5 天后,他们惊讶地宣布,计算速度已经超过了每秒 100 亿亿次(每秒 10^18 次),比起最尖端的一批超级计算机,它的运算速度高了一个数量级。这些运算能力被源源不断地投入到新冠病毒蛋白的相关研究中,尖刺蛋白 – ACE2 蛋白结合体、新冠病毒蛋白酶、抗体蛋白等模拟项目,正在不断地拆分发送给世界各地的参与者。计算资源永远不嫌多。除了勤洗手、戴口罩、尽量待在家里之外,只要有一台能上网的电脑,任何人都可以为抗击疫情做出进一步的贡献。前往 Folding@home 项目的官网,下载客户端,安装,启动程序 —— 此时,你的电脑便准备好接收任务,为特效药的研发提供基础信息了。
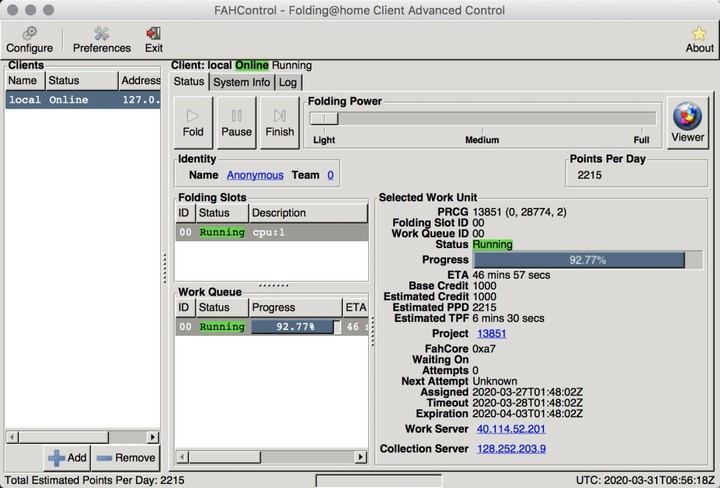
▲ Folding@home 的运行界面。可以在官网上查询到,此时运行的任务(PRCG:13851)是一个与 SARS-CoV 和 SARS-CoV-2 相关的研究 | FAHControl
除此之外,在伯克利大学网络计算开放平台(BOINC)上,生物医学项目 Rosetta@home 也在将重心调整到新冠病毒相关计算上,你甚至可以在智能手机上运行它。新冠疫情可能是 1918 年大流感以来最严重的瘟疫。这次,人类的手中握着与病魔抗争的崭新武器,它曾被用来凝视无尽的星空,如今它将帮助我们,拯救仰望同一片星空的生命。更重要的是,即便是完全不懂医学和生物学的普通人,也可以为挥舞这把利刃贡献一点点力量。而我们毕竟是数十亿人,相信这力量足以披荆斩棘。
参考文献
[1]Cruz MA, Frederick TE, Singh S, Vithani N, Zimmerman MI, Porter JR, Moeder KE, Amarasinghe GK, Bowman GR. 2020. Discovery of a cryptic allosteric site in Ebola’s ‘undruggable’ VP35 protein using simulations and experiments. bioRxiv:2020.02.09.940510.
[2]https://setiathome.berkeley.edu/
[3]https://www.theatlantic.com/science/archive/2017/05/aliens-on-your-packard-bell/527445/
[4]https://foldingathome.org/2020/02/27/foldinghome-takes-up-the-fight-against-covid-19-2019-ncov/
[5]https://foldingathome.org/2020/03/15/coronavirus-what-were-doing-and-how-you-can-help-in-simple-terms/
[6]https://www.dezeen.com/2020/03/25/coronavirus-cure-folding-at-home-software/
[7]https://medicine.wustl.edu/news/crowdsourced-supercomputing-project-sets-sights-on-coronavirus/
[8]http://boinc.bakerlab.org/rosetta/rah/rah_about.php